
14 de desembre de 2021 - Notes de Premsa
Aquest nou recurs, desenvolupat per l'equip del Grup de descobriment de fàrmacs basats en GPCRs de l'Institut Hospital del Mar d'Investigacions Mèdiques, permet analitzar en tres dimensions els moviments de les proteïnes que formen part del virus que provoca la COVID-19. Això pot ajudar als investigadors a entendre el seu funcionament i a desenvolupar nous tractaments i vacunes. L'eina, disponible en línia per a tots els investigadors, proporciona un gran nombre de simulacions del funcionament d'aquestes proteïnes, així com recursos per predir com pot canviar la seva funció en relació amb les mutacions que es poden produir en l'estructura d'aquest coronavirus. Els impulsors de la iniciativa han utilitzat més de 360 gigabits de dades per portar-la a terme. És l'única base de dades creada fins ara que combina simulacions de proteïnes amb dades de mutacions per estudiar el SARS-CoV-2.
Els investigadors disposen d'una nova eina per fer front al SARS-CoV-2, analitzar-lo i buscar noves formes per lluitar contra la COVID-19. Es tracta de la base de dades SCoV2-MD (que es pot consultar a www.scov2-md.org) que conté informació detallada, a nivell atòmic, dinàmic i en tres dimensions, de totes les proteïnes amb estructura tridimensional coneguda d'aquest coronavirus. En total, conté 360 gigabits de dades sobre la majoria de les 29 proteïnes que en formen part: quatre d'estructurals, setze de no-estructurals i nou d'accessòries. Nucleic Acids Research ha publicat un article sobre el seu funcionament, que ha considerat un dels més destacats entre els articles publicats a la revista.

Jana Selent i Mariona Torrens-Fontanals
Les proteïnes són molècules bàsiques en el funcionament de les cèl·lules. En el cas del SARS-CoV-2, el coronavirus responsable de la COVID-19, són les responsables de la seva capacitat d'infectar els éssers humans, així com de la seva propagació. És el cas de l'anomenada proteïna espiga, que forma la característica corona que dona el seu nom a aquest tipus de virus. La nova base de dades, impulsada pel Grup de descobriment de fàrmacs basats en GPCRs de l'Institut Hospital del Mar d'Investigacions Mèdiques (IMIM-Hospital del Mar), en col·laboració amb el Biophysics Institute (CNR-IBF) del National Research Council d'Itàlia, el Paul Scherrer Institute de Suïssa i Dompé Farmaceutici d'Itàlia, permet conèixer amb un grau de detall mai vist la seva estructura i el seu funcionament, així com predir la seva evolució al llarg de les diferents mutacions que ha patit i que patirà el virus.
Aquesta eina, una de les més potents creades fins ara combinant simulacions de l'estructura tridimensional de les proteïnes amb dades de mutacions en el virus responsable de la COVID-19, està disponible per a qualsevol investigador, en línia i de forma gratuïta. En connectar-se, sense necessitat d'utilitzar cap aplicació específica, poden veure l'estructura de les proteïnes del SARS-CoV-2 a nivell atòmic, gràcies a l'anàlisi realitzat pels creadors de la base de dades a partir de la informació generada per ells mateixos i la disponible a diferents repositoris públics. Per fer-ho, s'han fet servir eines computacionals de simulació de la dinàmica molecular, que permeten fer prediccions de com cada àtom de la proteïna actuarà seguint les lleis de la física, per a construir així un model tridimensional del comportament d'aquesta molècula. Les simulacions acumulades, 252 en total, també preveuen l'impacte de les mutacions conegudes del coronavirus sobre les proteïnes.
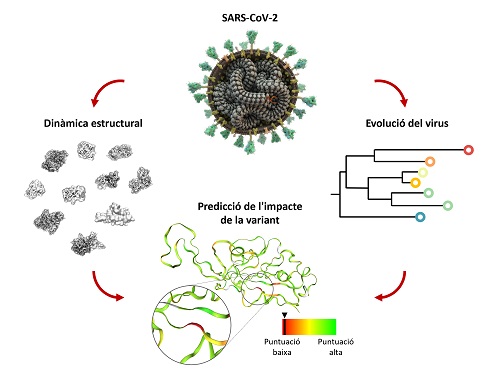
Imatge de la predicció de l’impacte d’una variant del SARS-CoV-2
"El que realment és nou és que utilitzem dades dinàmiques combinades amb l'evolució del virus per predir el seu impacte en la funció de les proteïnes", explica Jana Selent, investigadora de l'IMIM-Hospital del Mar i autora principal de l'article sobre aquesta nova eina. Per a fer-ho, "a banda de les simulacions del comportament de les proteïnes, s'ha tingut en compte la informació disponible sobre la genètica del virus per calcular i predir l'impacte de les mutacions", afegeix Toni Giorgino, coautor principal de l'article. Per aquest motiu, SCoV2-MD es converteix en una eina útil per visualitzar com aquestes mutacions del SARS-CoV-2 afectaran la seva capacitat de transmissió i per a infectar les cèl·lules humanes a través dels canvis que produeixen a les proteïnes que en formen part.
A la vegada, aquesta nova eina també pot ajudar als investigadors a desenvolupar nous tractaments i vacunes contra la COVID-19. "Veure les simulacions permet veure i entendre com es comporta, com funciona i quines parts de l'estructura de la proteïna són importants i possibles dianes per a l'estudi de nous tractaments", apunta Mariona Torrens-Fontanals, també investigadora de l'IMIM-Hospital del Mar i primera signant del treball. Fins i tot, facilita la possibilitat de predir si les mutacions del coronavirus poden afectar la capacitat dels anticossos que formen les vacunes contra la COVID-19 per reconèixer el virus i activar el sistema immunitari contra ell.
Mariona Torrens-Fontanals, Alejandro Peralta-García, Carmine Talarico, Ramon Guixà-González, Toni Giorgino, Jana Selent, SCoV2-MD: a database for the dynamics of the SARS-CoV-2 proteome and variant impact predictions, Nucleic Acids Research, 2021; https://doi.org/10.1093/nar/gkab977
En combinar informació de la dinàmica estructural de proteïnes i dades de l'evolució del virus, SCoV2-MD permet estudiar l'impacte de mutacions específiques (per exemple, T24A a la proteasa principal SARS-CoV-2) en la unió de compostos antivirals (per exemple, l'inhibidor ML188).
Parc Salut Mar
Passeig Marítim 25-29 Barcelona 08003
Vegeu la situació a Google Maps
Tel: 93 248 30 00 · Fax: 93 248 32 54
Sol·licitud d'informació
© 2006 - 2024 Parc de Salut Mar · Avís Legal i Privacitat de dades | Política de Cookies | Accessibilitat